Síntese proteica: a produção de proteínas – Resumo
Entenda o que é tradução e como as proteínas são sintetizadas
A síntese proteica consome mais energia da célula do que qualquer outro processo metabólico. Por sua vez, as proteínas representam mais massa do que qualquer outro componente de organismos vivos (com exceção da água) e as proteínas desempenham praticamente todas as funções de uma célula.
Nessa aula veremos o passo a passo de como uma célula sintetiza suas proteínas, as fases e estruturas envolvidas no processo de produção de proteínas.
O que é síntese proteica
Síntese proteica é o processo realizado pelos ribossomos para produzir proteínas a partir de moléculas de RNA. É por meio desse processo que as células sintetizam suas proteínas. A produção de proteínas pode ser resumida em três fases: transcrição, ativação e tradução.
Como ocorre a produção de proteínas
O processo de tradução, ou síntese proteica, envolve a decodificação de uma mensagem contida no RNA mensageiro (mRNA) para um produto polipeptídico.
Os aminoácidos são covalentemente encadernados entre si por interligação de ligações peptídicas em comprimentos variando de aproximadamente 50 resíduos de aminoácidos a mais de 1.000.
Cada aminoácido indivíduo tem um grupo amino (NH₂) e um grupo carboxilo (COOH). Os polipéptidos são formados quando o grupo amino de um aminoácido forma uma ligação amida (isto é, péptido) com o grupo carboxilo de outro aminoácido. Esta reação é catalisada por ribossomos e gera uma molécula de água.
Uma ligação peptídica liga a extremidade carboxilo de um aminoácido com a extremidade amino de outro, expulsando uma molécula de água.
Para simplificar nesta imagem, apenas os grupos funcionais envolvidos na ligação peptídica são mostrados. As designações R e R referem-se ao resto de cada estrutura de aminoácidos.
Uma máquina de sintetizar proteína
Além do modelo de mRNA, muitas moléculas e macromoléculas contribuem para o processo de tradução. A composição de cada componente pode variar entre as espécies; por exemplo, os ribossomos podem consistir em diferentes números de RNA ribossômico (rRNA) e polipéptidos dependendo do organismo.
No entanto, as estruturas e funções gerais da maquinaria de síntese proteica são comparáveis de bactérias a células humanas. A tradução requer a entrada de um modelo de mRNA, ribossomos, tRNAs e vários fatores enzimáticos.
Mesmo antes de um mRNA ser traduzido, uma célula deve investir energia para construir cada um dos seus ribossomos.
Em E. coli , existem entre 10.000 e 70.000 ribossomos presentes em cada célula em qualquer momento.
Afinal, o que é um ribossomo
Um ribossomo é uma macromolécula complexa composta por rRNA estruturais, catalíticos e muitos polipéptidos distintos. Em eucariotas, o nucléolo é completamente especializado para a síntese e montagem de rRNAs.
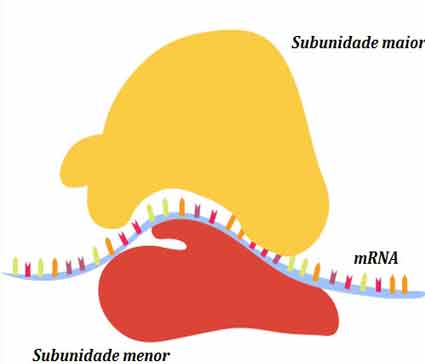
Os ribossomos existem no citoplasma nos procariotos e no citoplasma e retículo endoplasmático áspero em eucariotas.
As mitocôndrias e os cloroplastos também possuem seus próprios ribossomos na matriz e no estroma, que parecem mais parecidos com os ribossomos procarióticos (e têm sensibilidade similar aos fármacos) do que os ribossomos que se encontram fora das suas membranas externas no citoplasma.
Os ribossomos se dissociam em subunidades grandes e pequenas quando não estão sintetizando proteínas e se associam durante o início da tradução.
Em E. coli, a subunidade pequena é descrita como 30S, e a subunidade grande é 50S, para um total de 70S (lembre-se de que as unidades Svedberg não são aditivos).
Os ribossomos de mamíferos têm uma pequena subunidade 40S e uma grande subunidade 60S, para um total de 80S. A subunidade pequena é responsável por ligar o modelo de mRNA, enquanto a subunidade grande se liga sequencialmente o tRNAs.
Cada molécula de mRNA é traduzida simultaneamente por muitos ribossomos, todas as proteínas de síntese na mesma direção: leitura do mRNA de 5 ‘a 3’ e síntese do polipéptido do terminal N ao terminal C. A estrutura completa de mRNA / poli-ribossomos é chamada de polissomo como na imagem abaixo.
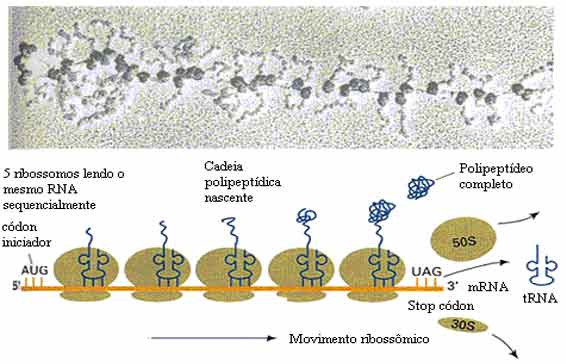
tRNAs: os RNAs transportadores
Os RNA transportadores (RNAt) são moléculas de RNA estrutural que foram transcritas a partir de genes pela RNA polimerase III.
Dependendo da espécie, existem 40 a 60 tipos de RNAt no citoplasma. Servindo como adaptadores, tRNAs específicos se ligam a sequências no modelo de mRNA e adicionam o aminoácido correspondente à cadeia polipeptídica. Portanto, os tRNAs são as moléculas que realmente “traduzem” a linguagem do RNA na linguagem das proteínas.
Dos 64 codões de mRNA possíveis – ou as combinações de três nucleotídeos de A, U, G e C-3 especificam o término da síntese proteica e 61 especificam a adição de aminoácidos à cadeia de polipéptidos. Destes 61, um codão (AUG) também codifica o início da tradução.
Cada anticódon do tRNA pode basear par com um dos codões de mRNA e adicionar uma tradução de aminoácidos ou terminar, de acordo com o código genético.
Por exemplo, se a sequência CUA ocorreu em um modelo de mRNA no quadro de leitura apropriado, ele iria ligar um tRNA que expressa a sequência complementar, GAU, que seria ligada ao aminoácido leucina.
Como as moléculas adaptadoras da tradução, é surpreendente que os tRNAs possam caber tanto especificidade em um pacote tão pequeno. Considere que os tRNAs precisam interagir com três fatores:
- devem ser reconhecidos pela aminoacil sintetase correta;
- devem ser reconhecidos pelos ribossomos; e
- devem se ligar à sequência correta no mRNA.
O aminoacil tRNA sintetase
O processo de síntese tRNA pela RNA polimerase III apenas cria uma porção de RNA da molécula adaptadora. O aminoácido correspondente deve ser adicionado mais tarde, uma vez que o tRNA é processado e exportado para o citoplasma.
Através do processo de tRNA “carregar”, cada molécula de tRNA está ligada ao seu aminoácido correto por um grupo de enzimas chamadas aminoacil tRNA sintetase.
Pelo menos um tipo de aminoacil tRNA sintetase existe para cada um dos 20 aminoácidos; o número exato de aminoácidos tRNA sintetases varia de acordo com as espécies.
Estas enzimas primeiro se ligam e hidrolisam ATP para catalisar uma ligação de alta energia entre um aminoácido e monofosfato de adenosina (AMP); uma molécula de pirofosfato é expulso nesta reação. O aminoácido ativado é então transferido para o RNAt, e o AMP é liberado.
O Mecanismo de síntese proteica
Tal como acontece com a síntese de mRNA, a síntese proteica pode ser dividida em três fases: iniciação, alongamento e término. O processo de tradução é semelhante em procariotas e eucariotas. Aqui vamos explorar como ocorre a tradução em E. coli , um procarioto representativo e especificar quaisquer diferenças entre a tradução procariótica e eucariótica.
Iniciação da Tradução
A síntese de proteínas começa com a formação de um complexo de iniciação. Em E. coli , este complexo envolve o pequeno ribossomo 30S, o modelo de mRNA, três fatores de iniciação (IFs, IF-1, IF-2 e IF-3), e um iniciador especial tRNA chamado tRNA.
O iniciador tRNA interage com o códon de início AUG (ou raramente, GUG), liga-se a uma metionina chamada fMet e também pode ligar IF-2.
No mRNA de E. coli , uma seqüência a montante do primeiro codão AUG, chamado de sequência Shine-Dalgarno (AGGAGG), interage com as moléculas de rRNA que compõem o ribossomo.
Essa interação âncora a subunidade ribossômica 30S no local correto no modelo de mRNA. O trifosfato de guanosina (GTP), que é um trifosfato de nucleótido de purina, atua como fonte de energia durante a tradução – tanto no início do alongamento quanto durante a translocação do ribossomo.
Em eucariotas, um complexo de iniciação semelhante forma, compreendendo mRNA, a subunidade ribossômica pequena 40S, IFs e nucleósidos trifosfatos (GTP e ATP). O iniciador carregado tRNA, chamado Met-tRNA i , não liga fMet em eucariotas, mas é distinto de outros Met-tRNAs na medida em que pode ligar FIs.
Tradução de proteínas
Nos procariotas e eucariotas, os fundamentos do alongamento são os mesmos, então analisaremos o alongamento na perspectiva de E. coli . A subunidade ribossomal 50S de E. coli consiste em três compartimentos: o sítio A (aminoacil) liga os tRNAs aminoácidos carregados recebidos.
O sítio P (peptidil) liga os tRNAs carregados que transportam aminoácidos que formam ligações peptídicas com a cadeia polipeptídica em crescimento, mas ainda não se dissociaram do seu tRNA correspondente.
O site E (saída) libera tRNAs dissociados para que possam ser recarregados com aminoácidos livres. Existe uma exceção a esta linha de montagem de tRNA.
Durante o alongamento da tradução, o modelo mRNA fornece especificidade. À medida que o ribossomo se move ao longo do mRNA, cada codão do mRNA entra em registro e é assegurada a ligação específica com o correspondente tRNA anticódon carregado. Se o mRNA não estivesse presente no complexo de alongamento, o ribossomo ligaria os RNAt de forma não específica.
O alongamento prossegue com tRNAs carregados entrando no site A e depois deslocando para o site P seguido pelo site E com cada “passo” de códon único do ribossomo.
Os passos ribossômicos são induzidos por mudanças conformacionais que avançam o ribossomo por três bases na direção 3 ‘. A energia para cada passo do ribossomo é doada por um fator de alongamento que hidrolisa GTP.
Ligações peptídicas formam-se entre o grupo amino do aminoácido ligado ao RNAt do sítio A e o grupo carboxilo do aminoácido ligado ao tRNA do site P.
A formação de cada ligação peptídica é catalisada pela peptidil transferase, uma enzima baseada em RNA que é integrada na subunidade ribossômica 50S.
A energia para cada formação de ligação peptídica é derivada da hidrólise de GTP, que é catalisada por um fator de alongamento separado.
O aminoácido ligado ao tRNA do site P também está ligado à cadeia polipeptídica em crescimento. À medida que o ribossomo passa através do mRNA, o antigo RNAt do P-site entra no site E, se destaca do aminoácido e é expulso.
Surpreendentemente, o aparelho de tradução de E. coli leva apenas 0,05 segundos para adicionar cada aminoácido, o que significa que uma proteína de 200 aminoácidos pode ser traduzida em apenas 10 segundos.
A terminação da tradução ocorre quando um codão sem sentido (UAA, UAG ou UGA) é encontrado. Após o alinhamento com o site A, esses códon sem sentido são reconhecidos por fatores de liberação em procariotas e eucariotas que indicam a peptidil transferase para adicionar uma molécula de água à extremidade carboxílica do aminoácido do site P. Esta reação força o aminoácido do site P para se separar do seu RNAt, e a proteína recém-produzida é liberada.
As subunidades ribossômicas pequenas e grandes se dissociam do mRNA e umas das outras; eles são recrutados quase imediatamente em outro complexo de iniciação da tradução. Depois que muitos ribossomos completaram a tradução, o mRNA é degradado para que os nucleotídeos possam ser reutilizados em outra reação de transcrição.
Enovelamento, modificação e segmentação
Durante e após a tradução, os aminoácidos individuais podem ser quimicamente modificados, as sequências de sinal podem ser anexadas e a nova proteína “se dobra” em uma estrutura tridimensional distinta como resultado de interações intramoleculares.
Uma sequência de sinal é uma queda curta de aminoácidos que direciona uma proteína para um compartimento celular específico.
Essas sequências na extremidade amino ou na extremidade carboxílica da proteína podem ser pensadas como o “bilhete de trem” da proteína para seu destino final.
Outros fatores celulares reconhecem cada sequência de sinal e ajudam a transportar a proteína do citoplasma para o compartimento correto.
Por exemplo, uma sequência específica no terminal amino direciona uma proteína para as mitocôndrias ou cloroplastos (em plantas). Uma vez que a proteína atinge seu destino celular, a sequência de sinal geralmente é cortada.
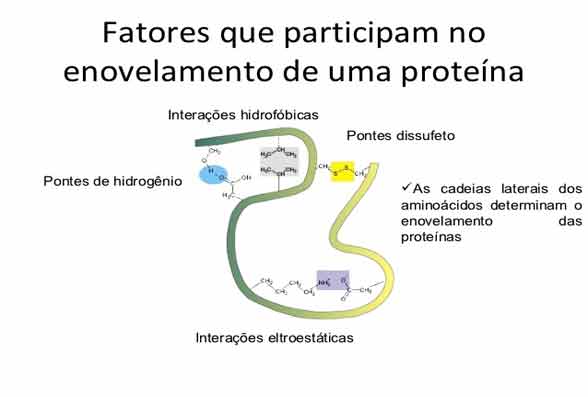
Muitas proteínas se dobram espontaneamente, mas algumas proteínas requerem moléculas auxiliares, chamadas chaperonas, para evitar que elas se agregam durante o complicado processo de dobramento.
Mesmo que uma proteína seja devidamente especificada pelo seu mRNA correspondente, pode assumir uma forma completamente disfuncional se as condições anormais de temperatura ou pH impedem que ele se dobre corretamente.
Resumo da aula Síntese proteica – Resumo
Os agentes durante a tradução incluem o de mRNA, ribossomos, tRNAs e vários fatores enzimáticos. A subunidade ribossômica pequena se forma no modelo de mRNA tanto na sequência de Shine-Dalgarno (procariotas e eucariotas). A tradução começa no AUG iniciante no mRNA, especificando metionina.
A formação de ligações peptídicas ocorre entre os aminoácidos sequenciais especificados pelo modelo mRNA de acordo com o código genético.
Os tRNAs carregados entram no sítio ribossômico A e os seus aminoácidos se unem com o aminoácido no local P. O mRNA inteiro é traduzido em “passos” de três nucleotídeos do ribossomo. Quando um códon sem sentido é encontrado, um fator de liberação liga e dissocia os componentes e libera a nova proteína. O dobramento da proteína ocorre durante e após a tradução.
Deixe seus comentários do que achou da aula Síntese proteica – Resumo.
Você sabia que tem bastante gente que envia posts, artigos, ou algo qualquer texto relacionado a ciências, educação ou biologia? Você gostaria de participar? Entre em contato.
Se você encontrar algo indevido nos textos, nas imagens, vídeos que recebemos, por favor, relate nos comentários que assim que puder será feita uma revisão.
Bibliografia da aula Síntese proteica – Resumo.
Youtube – síntese proteica – como as células produzem proteínas – Acesso em 17 de janeiro de 2021


ótima aula, fatam algumas imagens aí mas blz